Quickstart Hail (Korean)
스택 준비
- AWS CLI credential을 준비하고 터미널에서 적용합니다.
export AWS_DEFAULT_REGION="us-east-1"
export AWS_ACCESS_KEY_ID="{ACCESS_KEY}"
export AWS_SECRET_ACCESS_KEY="{SECRET_ACCESS_KEY}"
export AWS_SESSION_TOKEN="{SESSION_TOKEN}"- 이 CloudFormation 스택을 시작하려는 region에서 S3 버킷을 생성합니다.
이때 버킷이름은 자신의 이니셜을 사용해 만듭니다. 이미 동일한 버킷이 존재할 경우 생성할 수 없습니다.
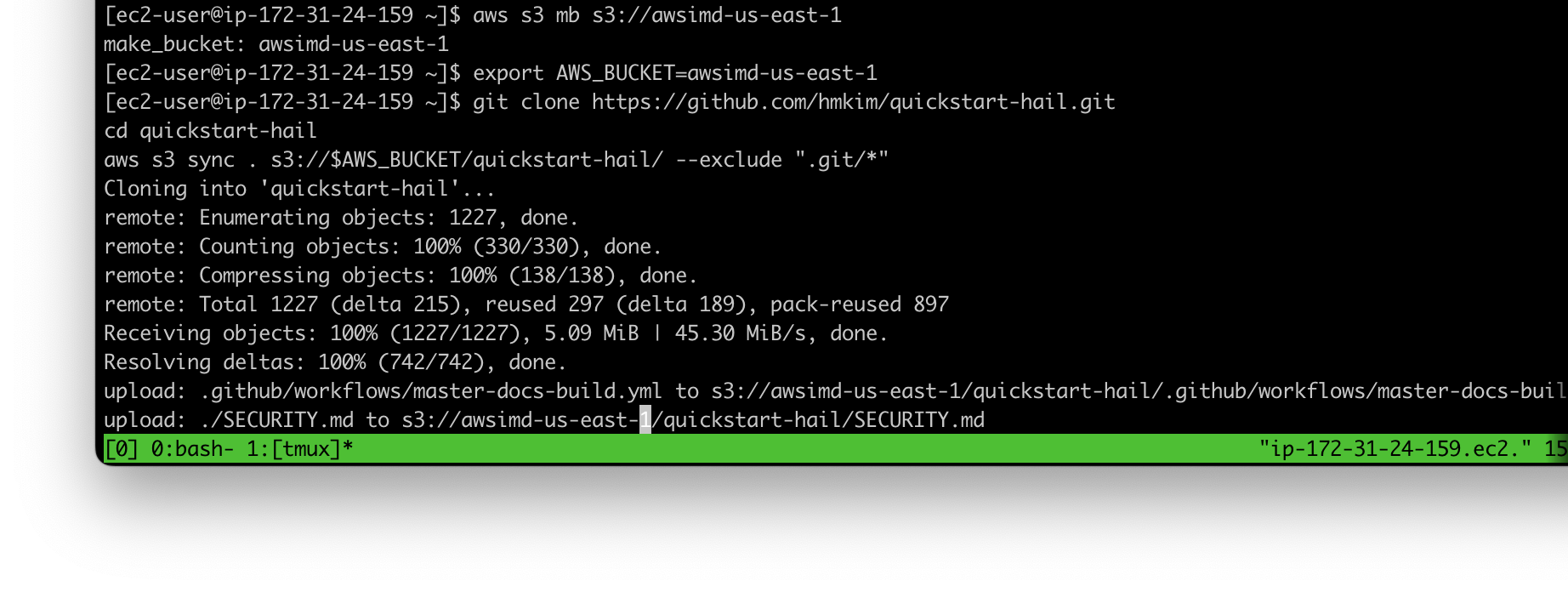
aws s3 mb s3://{버킷이름}-{리전}
이 리포지토리의 콘텐츠를 다운로드하고 압축을 푼 다음 다운로드한 콘텐츠를 앞에서 만든 S3 버킷에 넣습니다.
export AWS_BUCKET={버킷이름}-{리전}
git clone https://github.com/hmkim/quickstart-hail.git
cd quickstart-hail
aws s3 sync . s3://$AWS_BUCKET/quickstart-hail/ --exclude ".git/*"
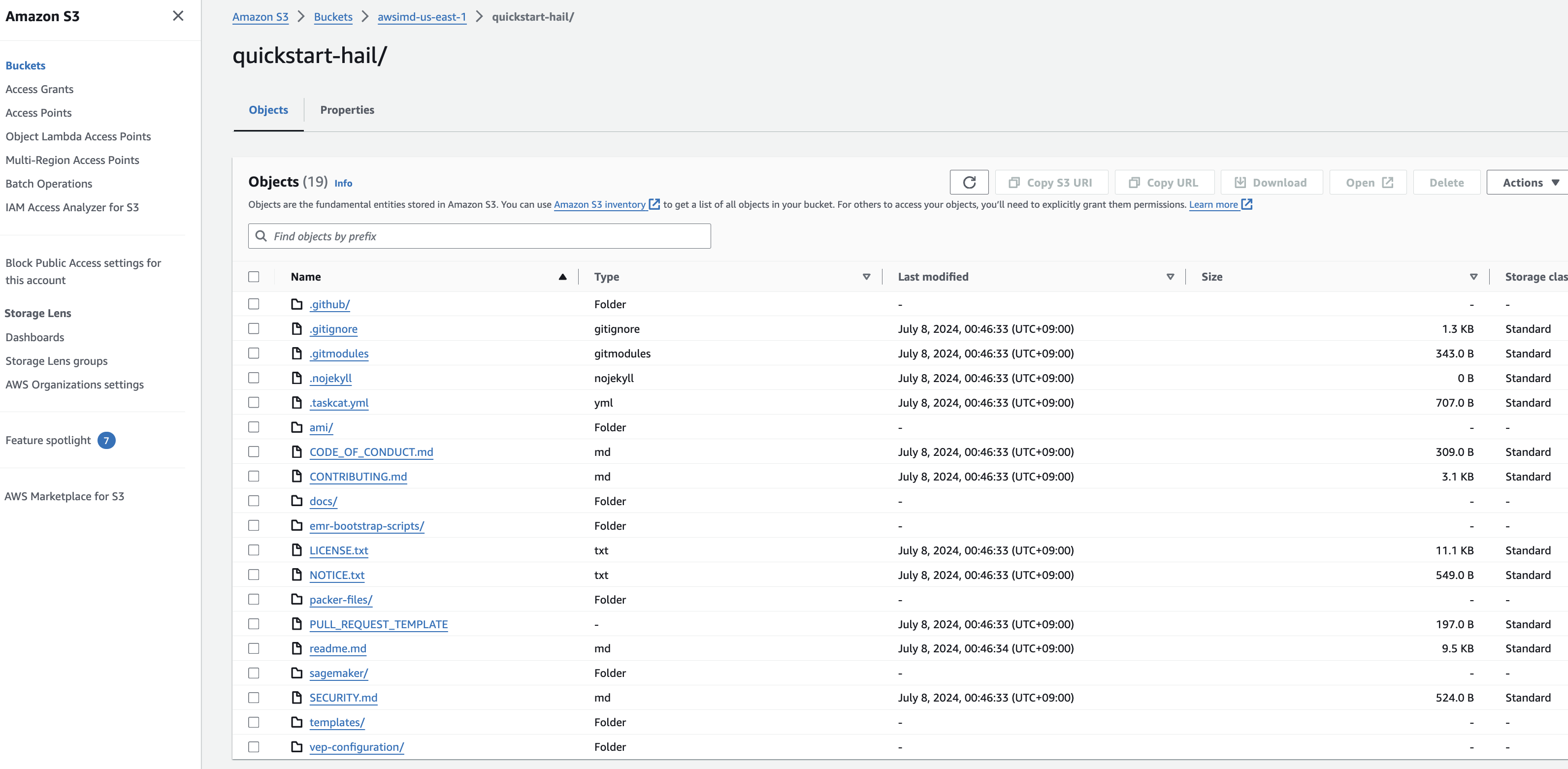
- Amazon S3 콘솔로 접속하여 버킷 및 디렉토리를확인합니다.
스택 실행
- CloudFormation 콘솔로 진입합니다.
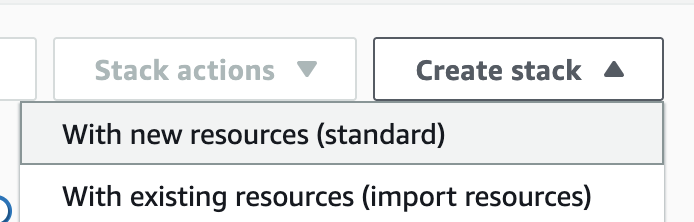
- 새로운 스택을 생성합니다. 이때 With new resources (standard)로 선택합니다.
- Amazon S3 콘솔로 접속하여 앞에서 업로드한 template 디렉토리 내의 hail-launcher.template.yaml을 선택하고 Copy URL을 클릭합니다. 경로는 다음과 같습니다.
{본인이만든버킷명} > quickstart-hail > templates > hail-launcher.template.yaml
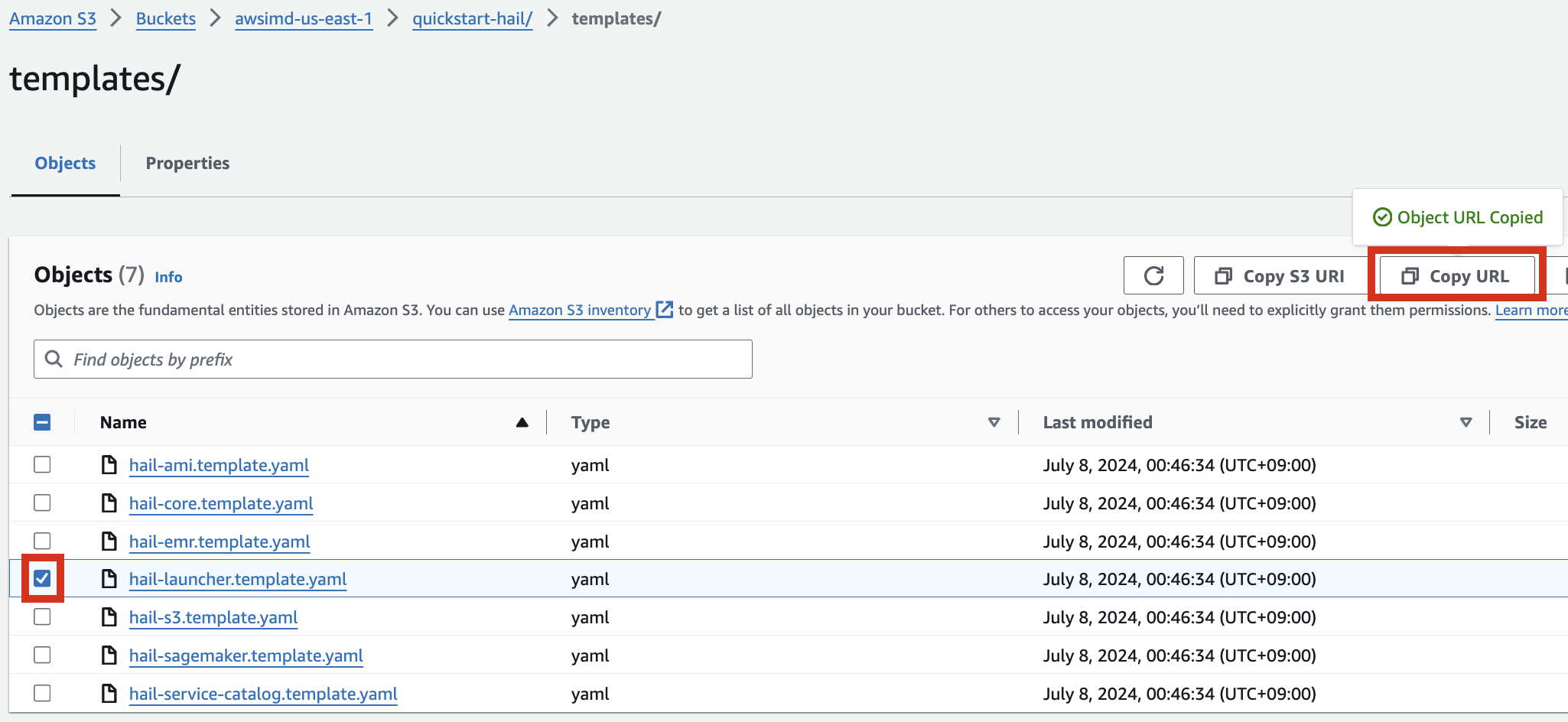
이 주소를 CloudFormation 스택 생성시 템플릿 주소로 입력하고 스택을 만듭니다.
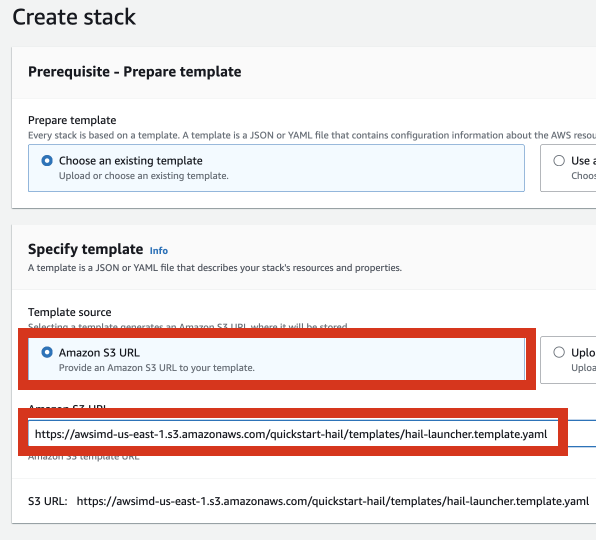
- Hail 스택을 만들기 위한 정보 입력을 진행합니다.
Stack 이름을 임의로 입력합니다.
VPC를 선택합니다. 같은 VPC내 Subnet을 하나 선택합니다. 이 실습에서는 public으로 선택합니다.

필요한 버킷들을 추가로 만들도록 설정해봅니다.
quickstart-hail 폴더를 업로드한 기존의 버킷명도 입력하고 리전도 확인합니다.
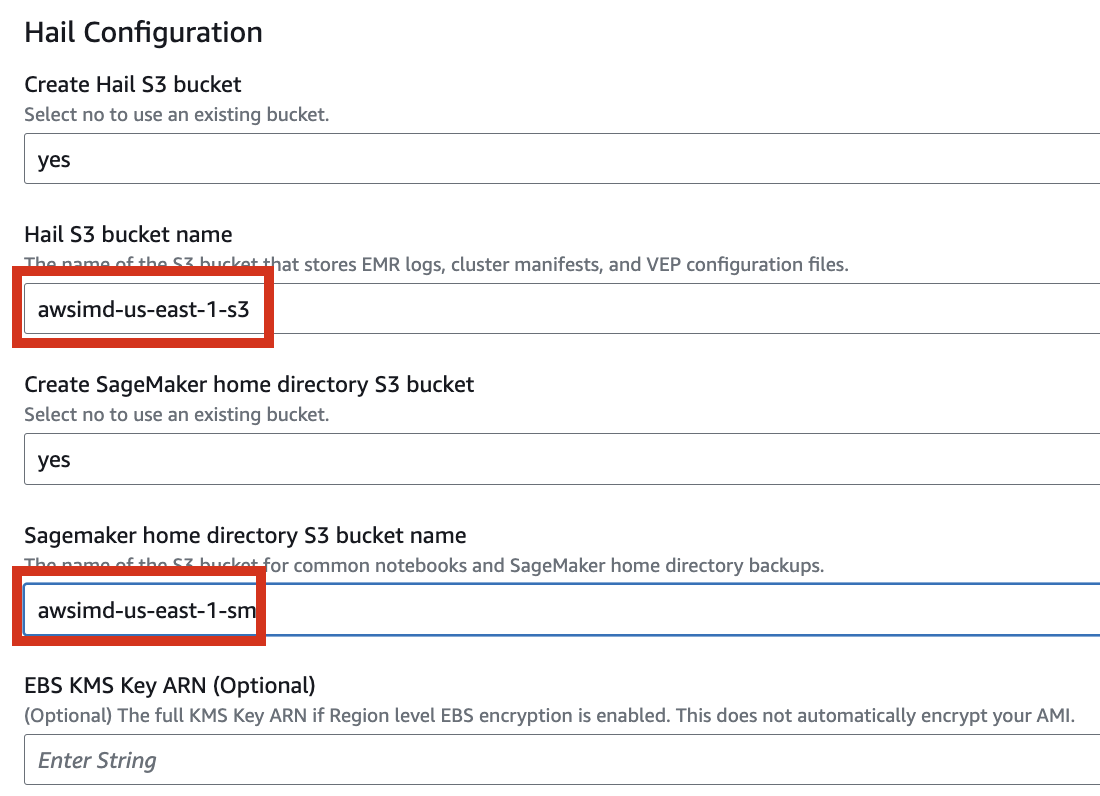
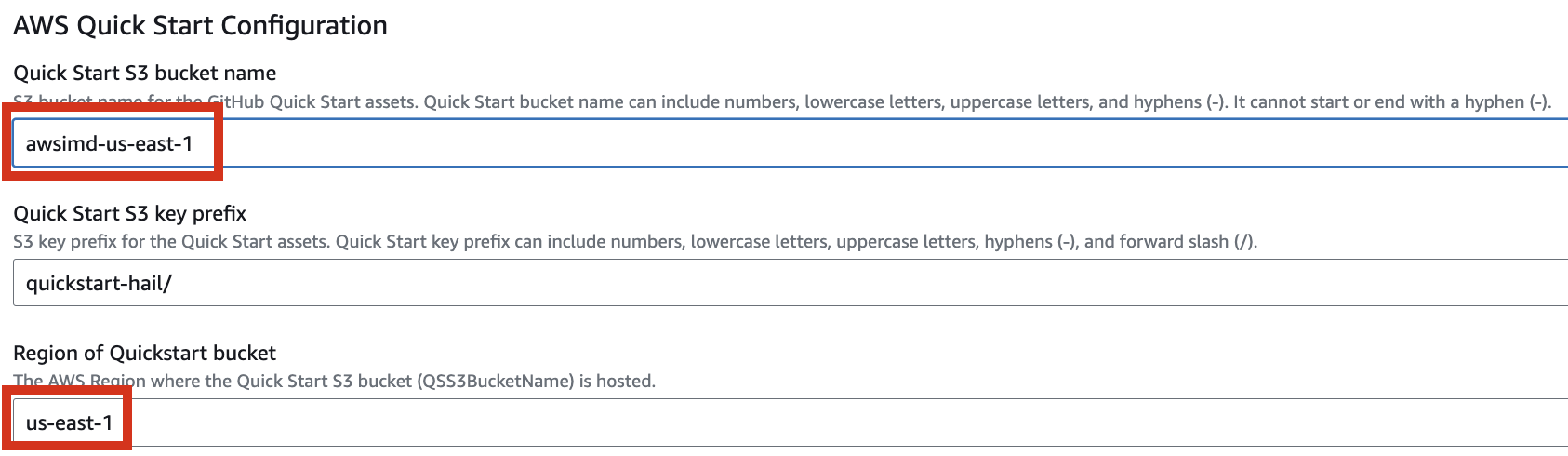
- 최종적으로 스택을 생성합니다.

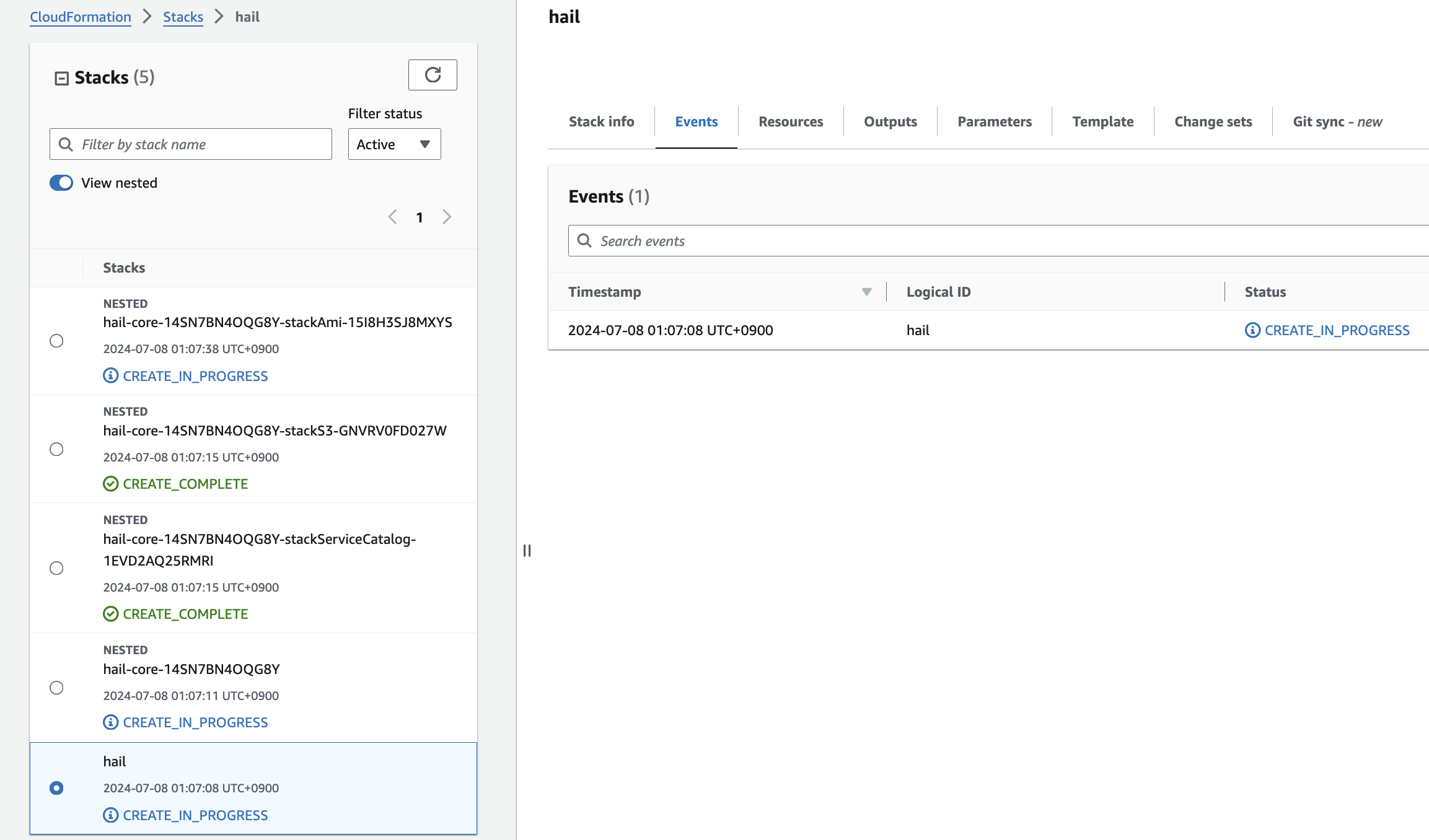
- CloudFormation 내에서 스택 생성을 확인합니다.
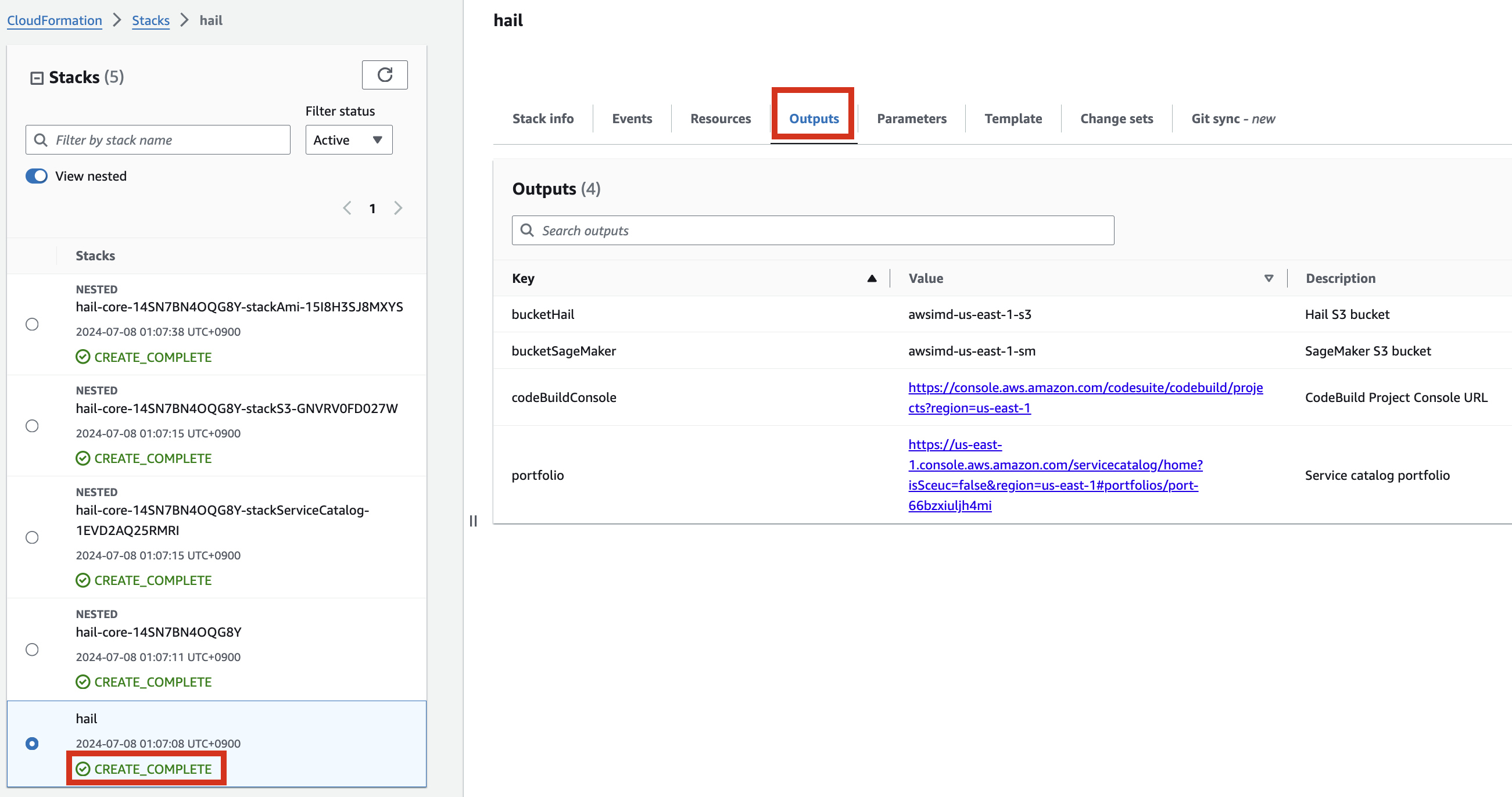
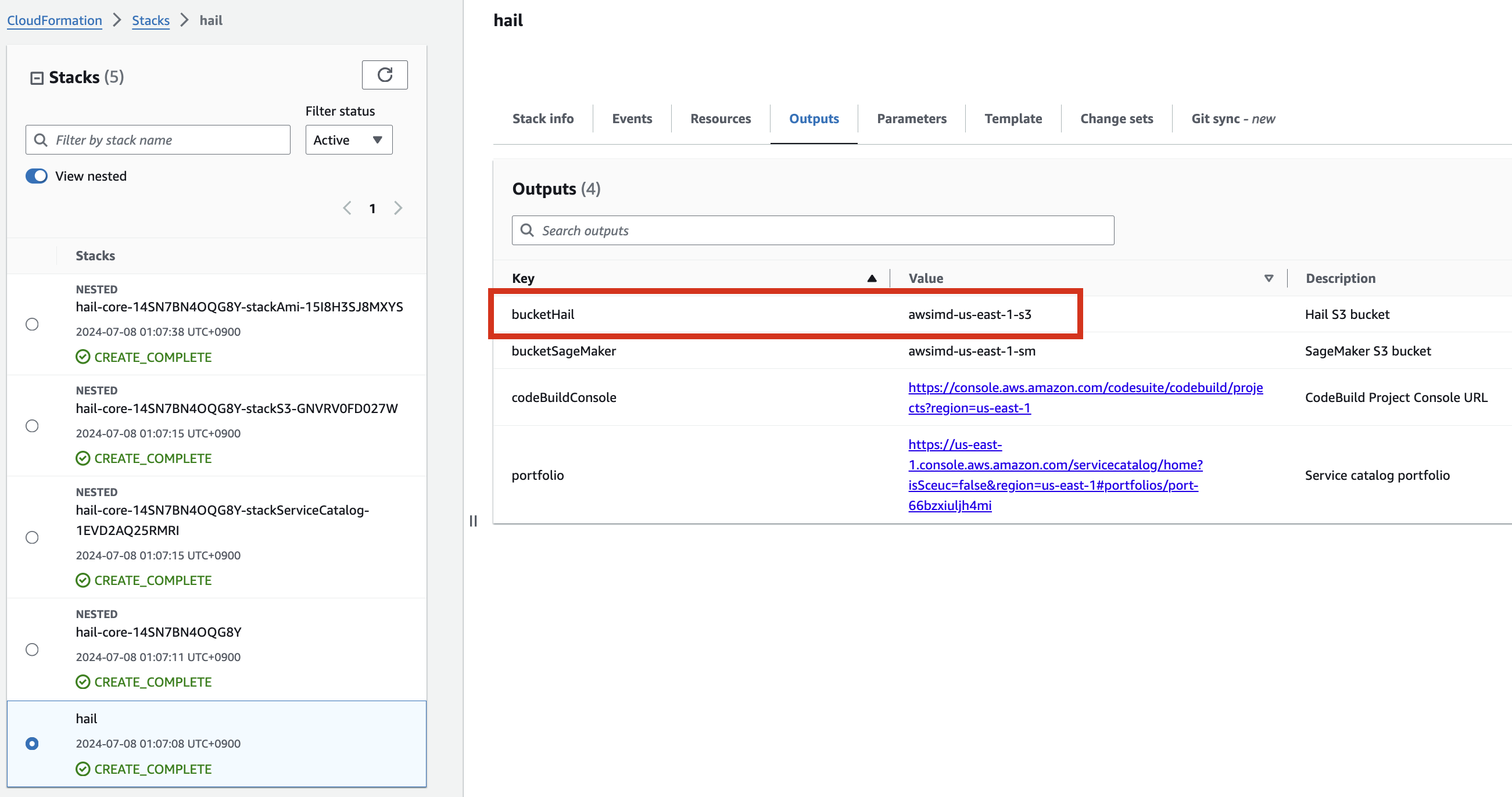
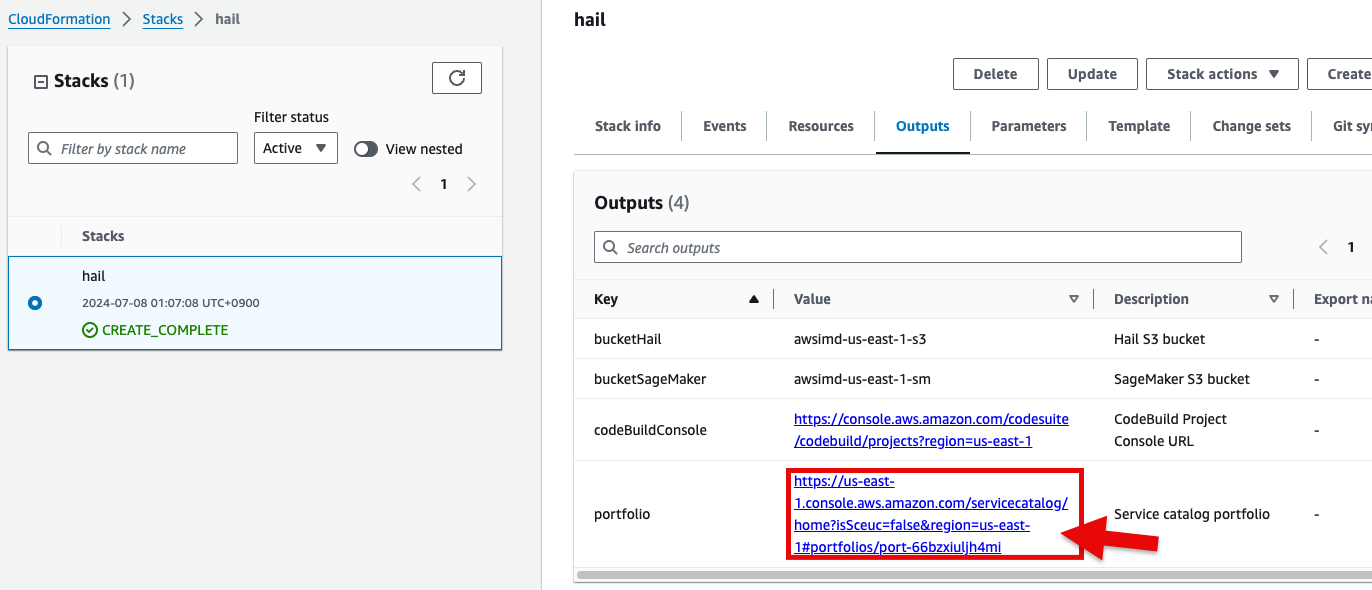
최상위 스택에서 CREATE_COMPLETE 메세지와 함께 아래와 같이 portfolio가 출력에 나왔다 면 정상 실행되었음을 확인할 수 있습니다.
Hail 및 VEP를 위한 AMI 생성
VEP 데이터 사전 다운로드 및 버킷 내 저장
VEP의 경우 미리 다운로드하여 앞에서 스택을 통해 생성 또는 입력한 버킷 (여기서는 CloudFormation의 Outputs 중 bucketHail 값을 사용했습니다.)에 위치시켜 놓을 수 있습니다.
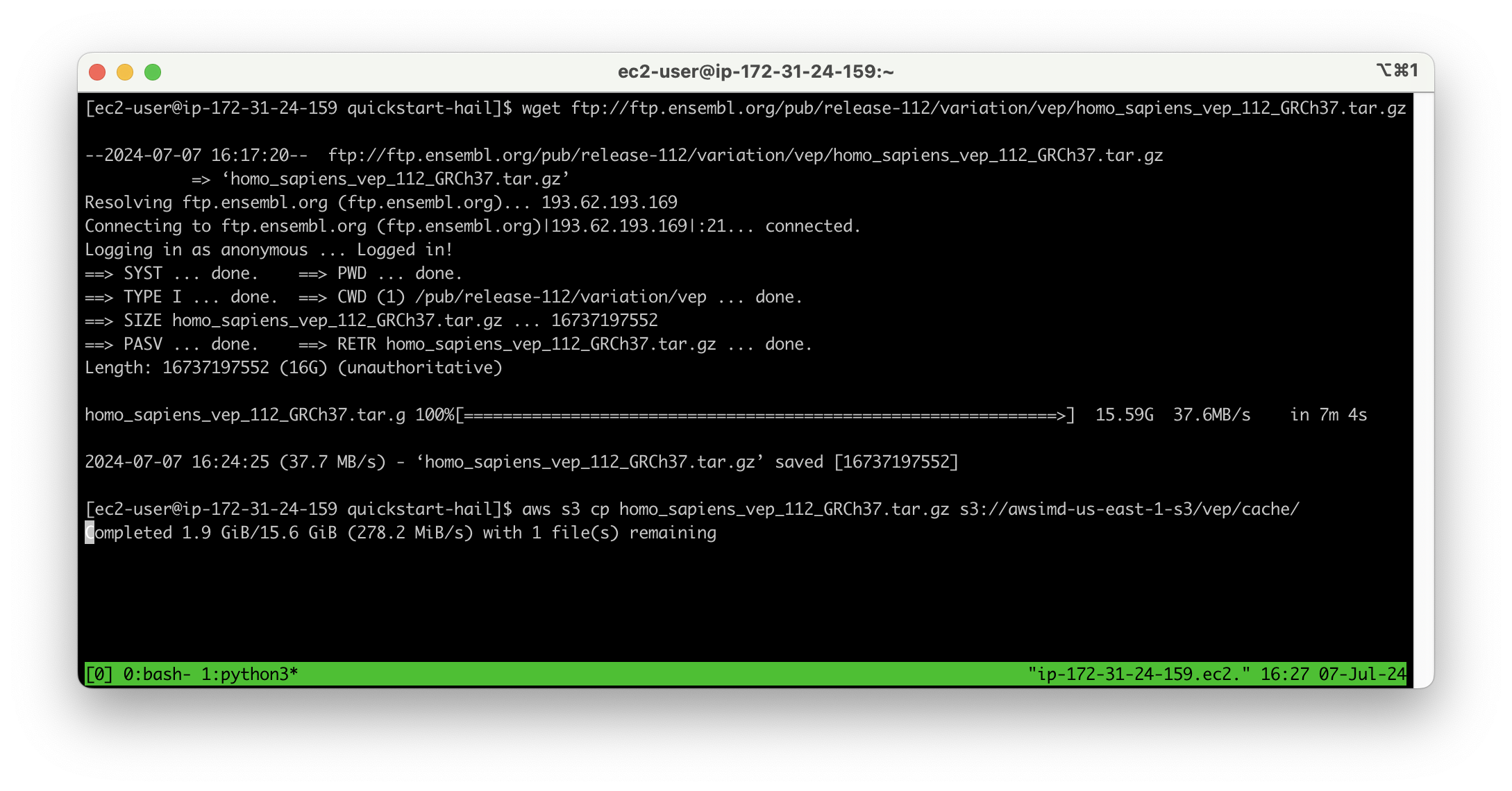
wget 명령어를 사용한 VEP 데이터 다운로드
wget ftp://ftp.ensembl.org/pub/release-112/variation/vep/homo_sapiens_vep_112_GRCh37.tar.gz
이후 버킷에 업로드합니다.
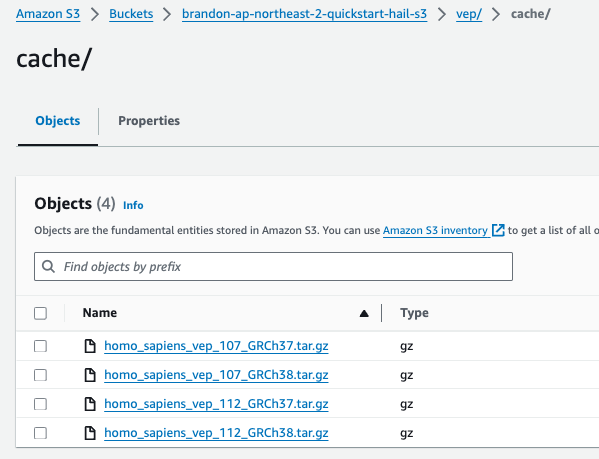
aws s3 cp homo_sapiens_vep_112_GRCh37.tar.gz s3://{버킷명}/vep/cache/
AMI 빌드
- CodeBuild 콘솔로 진입하여 각각 새로운 AMI 빌드를 시도합니다. Start build > Start with overrides를 선택합니다.
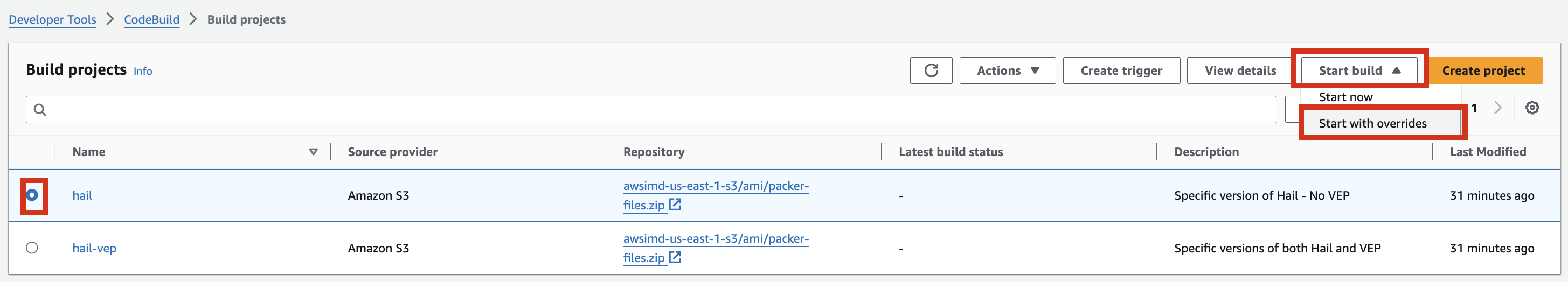
- Environment 섹션의 Additional configuration 을 확장해서 필요한 값을 입력합니다.
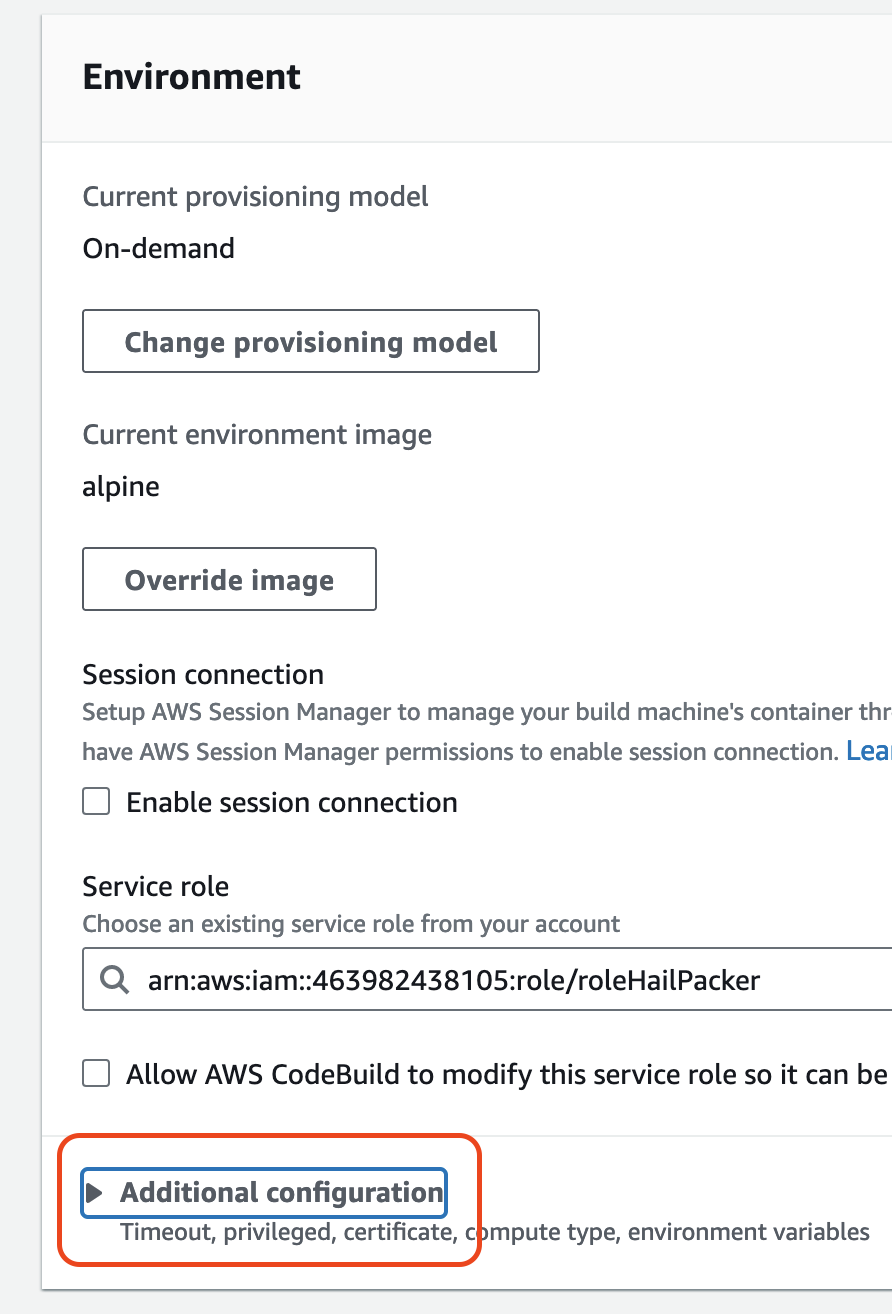
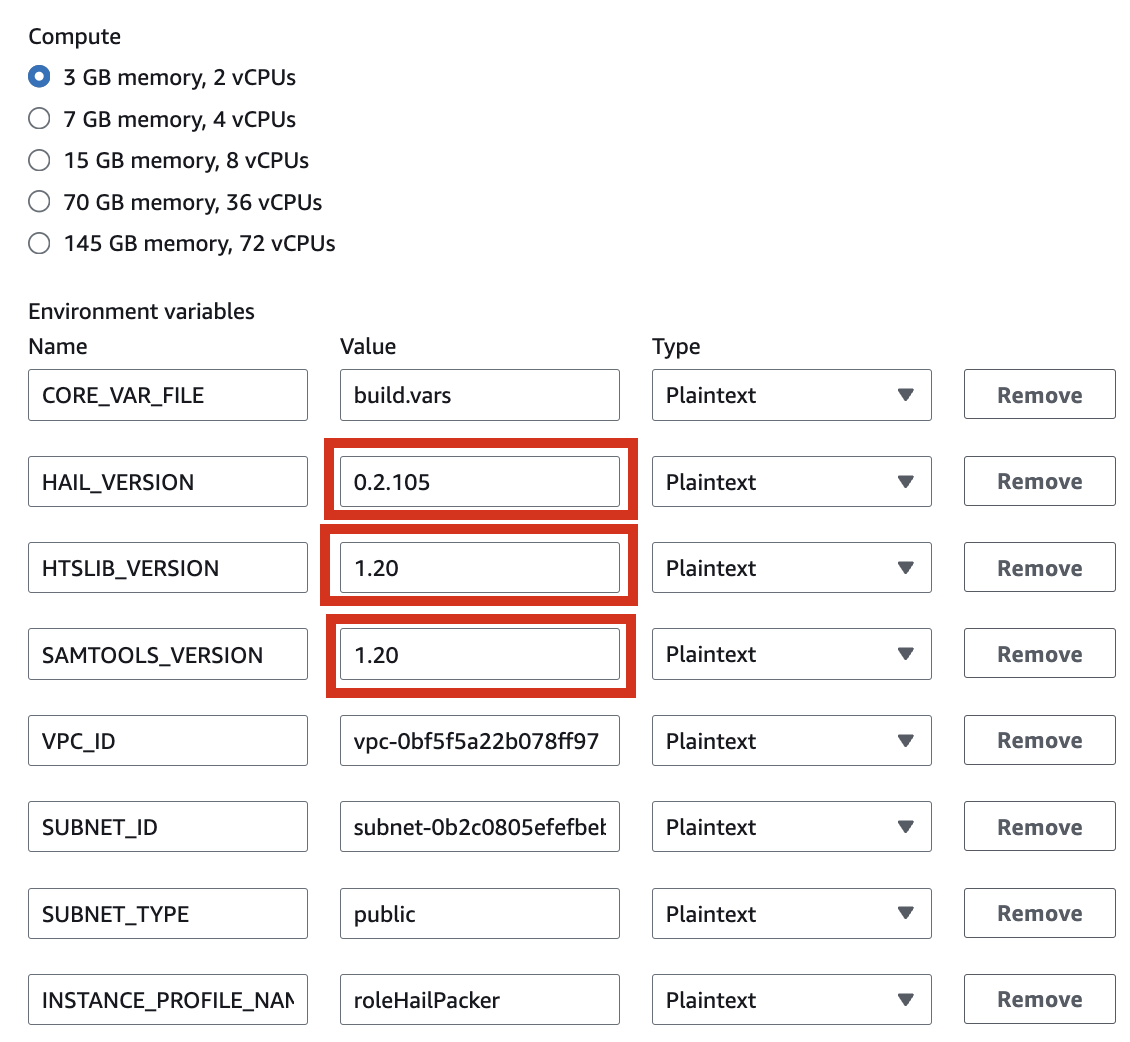
| HAIL_VERSION | 0.2.105 |
|---|---|
| HTSLIB_VERSION | 1.20 |
| SAMTOOLS_VERSION | 1.20 |
-
만일 hail-vep (VEP와 함께 설치) 옵션으로 빌드할 경우라면..
HAIL_VERSION 0.2.105 HTSLIB_VERSION 1.20 SAMTOOLS_VERSION 1.20 VEP_VERSION RODA_BUCKET <VEP 다운로드 받은 버킷명> 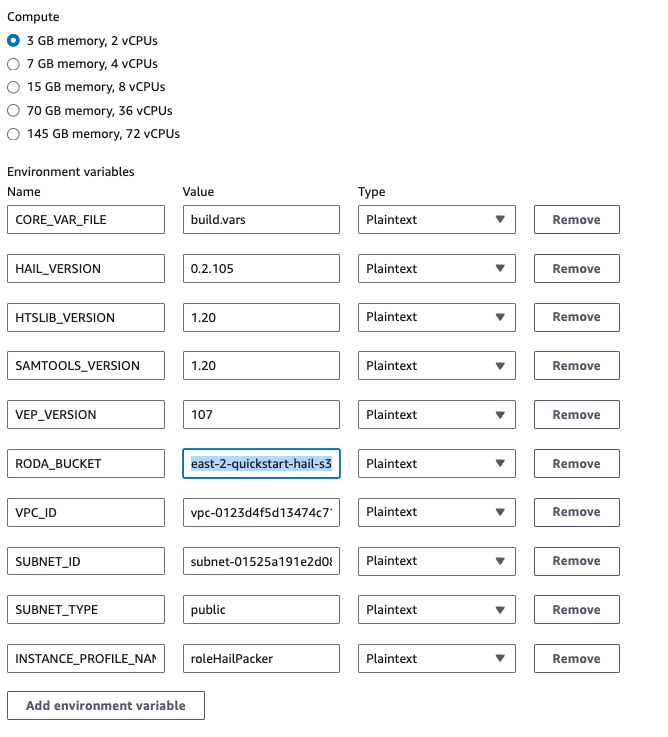
VEP 버전의 hail빌드시 약 1시간 38분 소요
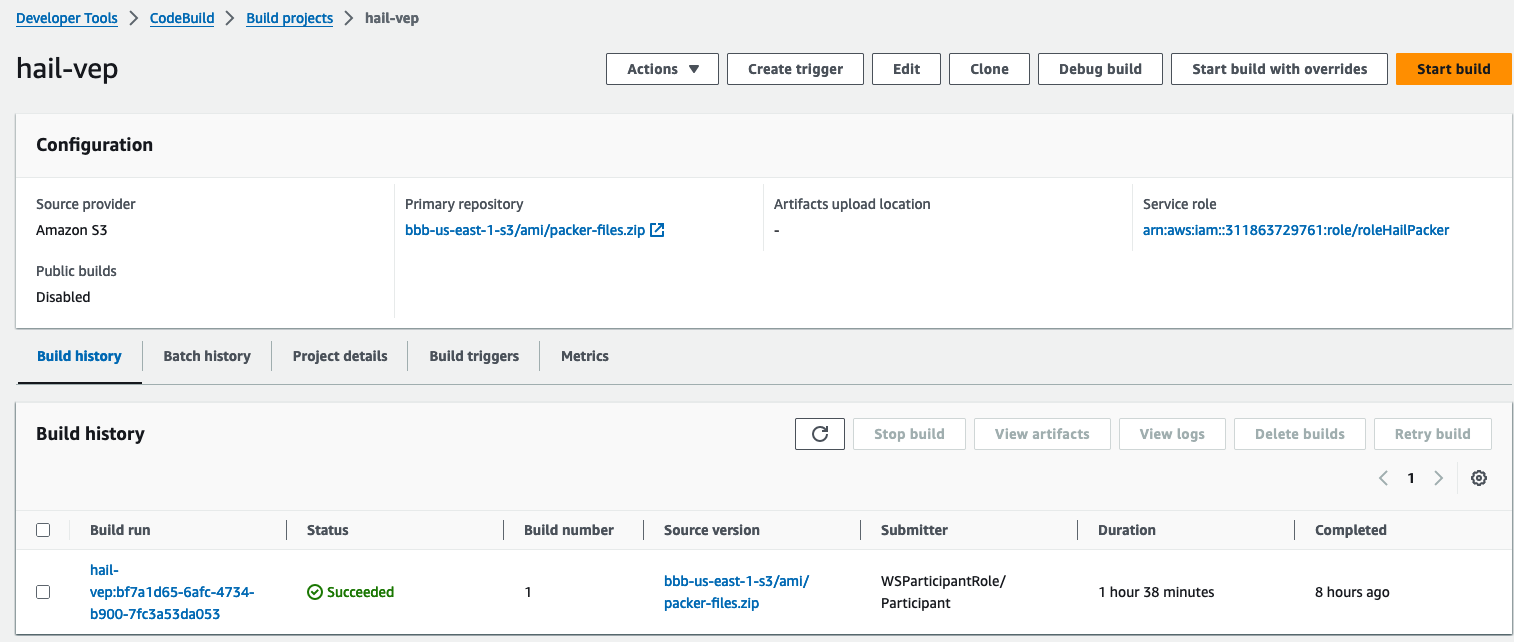
빌드 후 약 20분이 지나면 hail 이미지 빌드가 완료된 것을 확인할 수 있었습니다.
또한 AMI 결과는 AMI 메뉴 또는 CodeBuild 로그에서 확인 가능합니다.
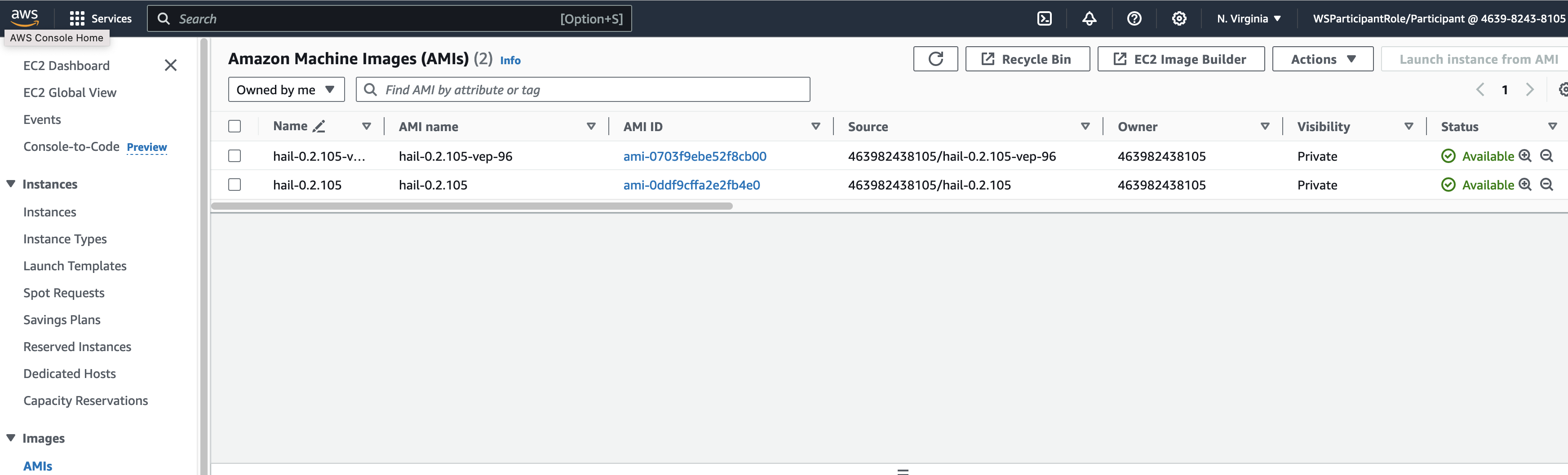
또는
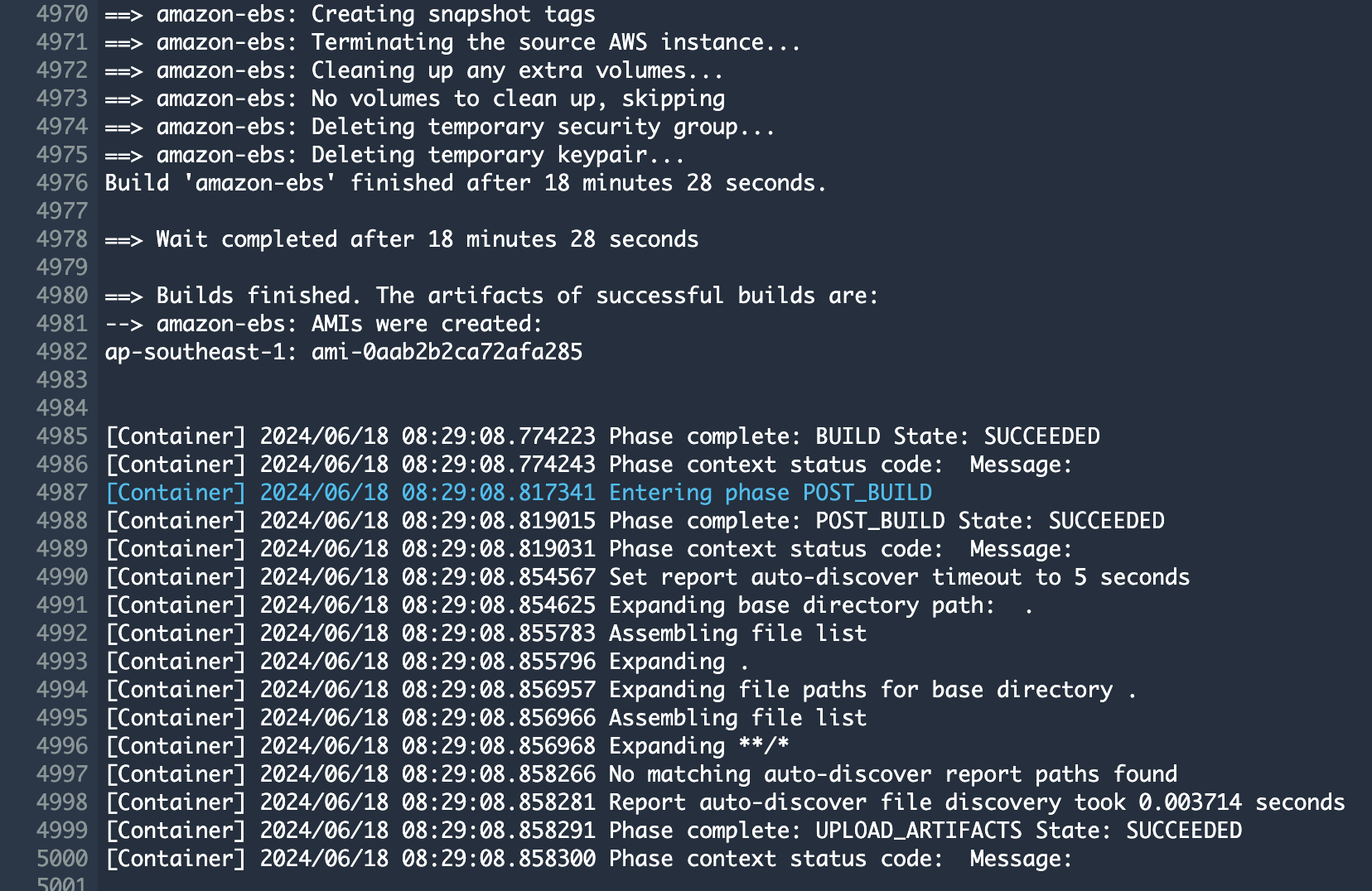
EMR 클러스터 실행 및 Jupyter 환경 세팅
EMR 클러스터 실행
- CloudFormation 서비스 콘솔에서 스택 Outputs 탭의 portfolio 에 있는 링크를 클릭합니다.
- 포트폴리오내 해당 Product에 대한 Access 탭을 클릭한 뒤 Grant access 를 클릭합니다.
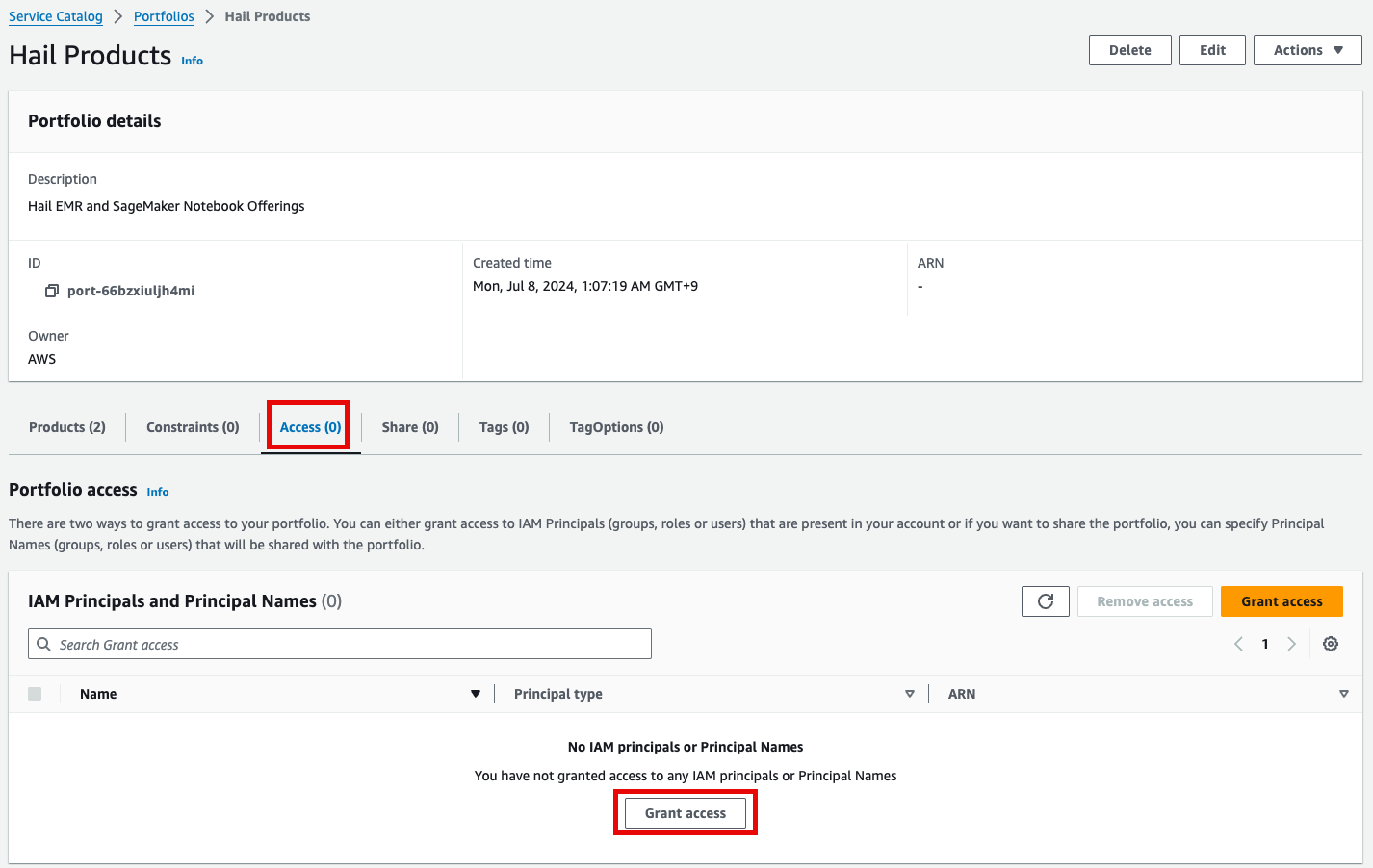
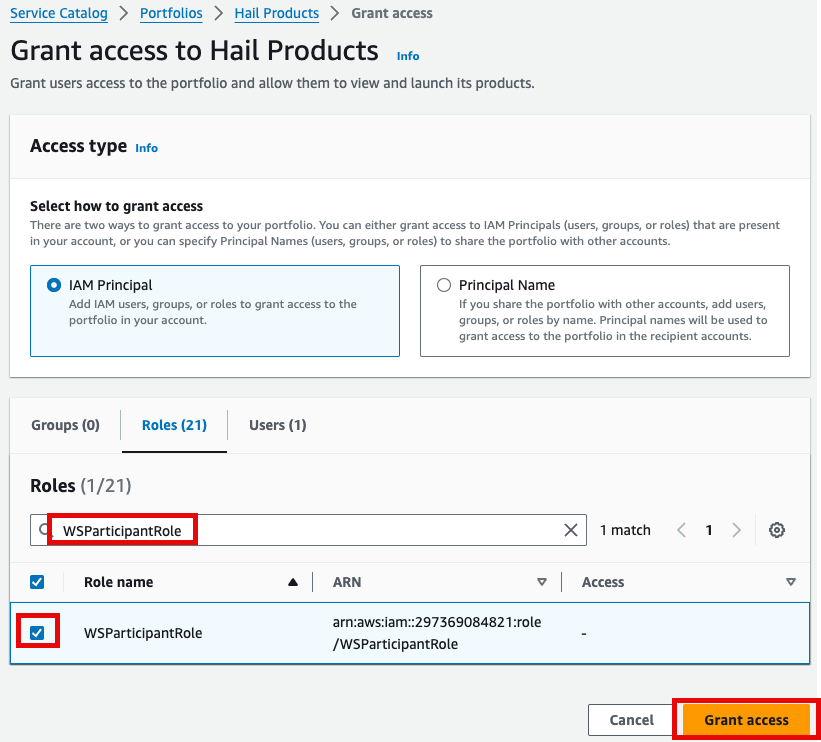
- 권한 추가를 합니다. 여기서는 실습 계정 역할 이름이
WSParticipantRole입니다. 검색후 체크하고 Grant access를 클릭합니다.
- Access 권한이 있음을 확인한 뒤 Provisioning 메뉴의 Product를 클릭하여 진입합니다.
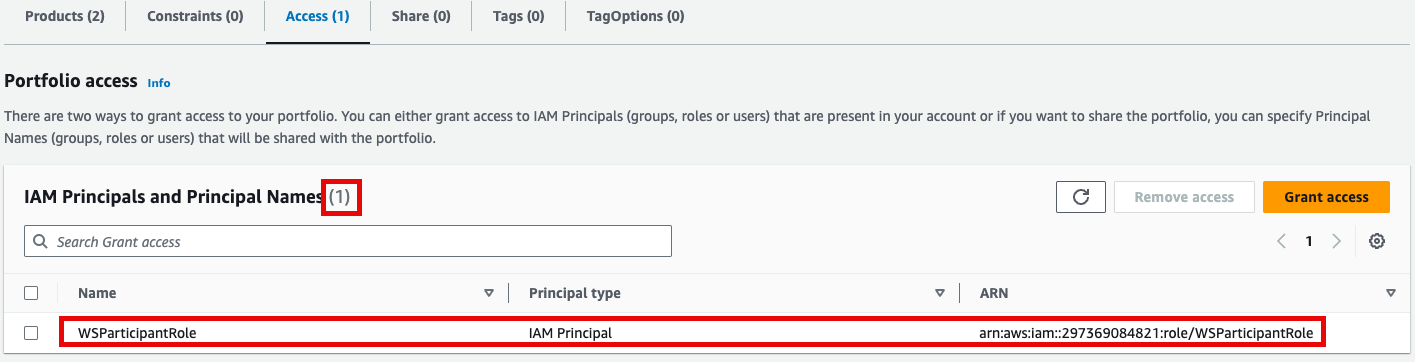
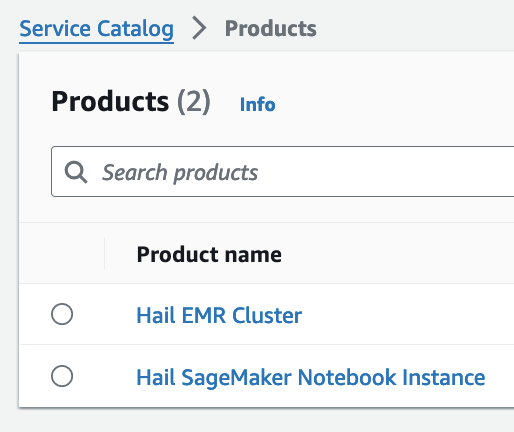
- 이제 권한이 있으므로 Products 항목에서 2개의 Product들을 볼 수 있게 되었습니다.
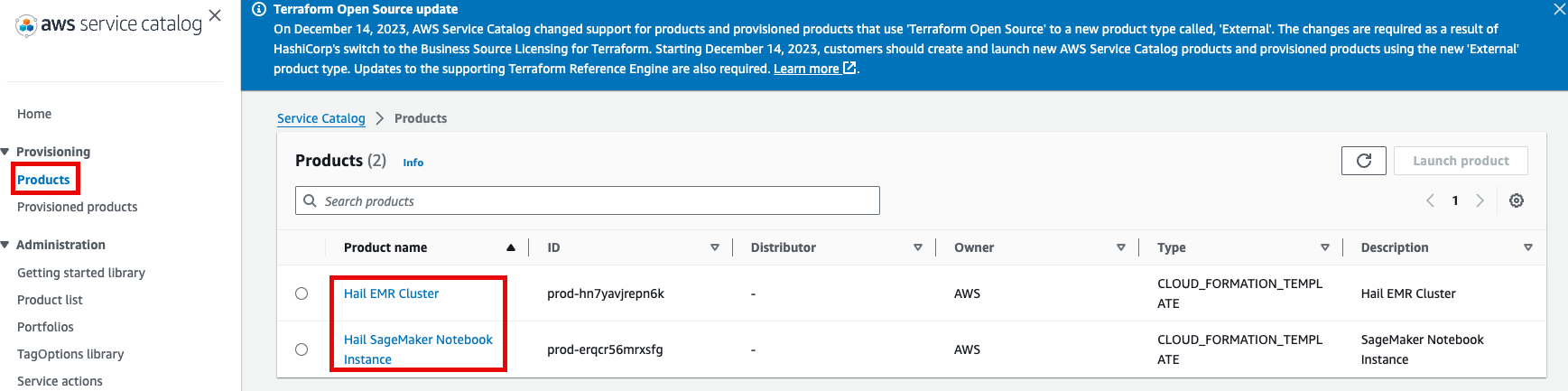
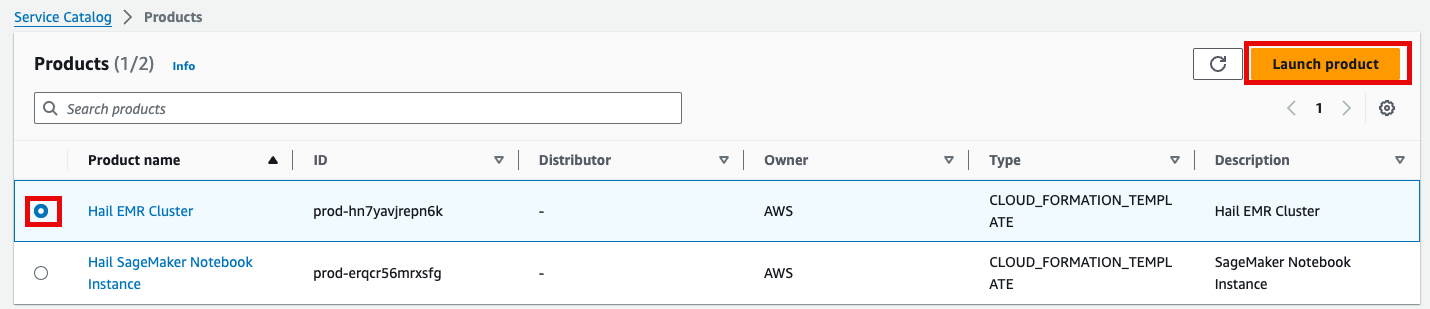
- Product에 있는 Hail EMR Cluster메뉴로 진입하여 원하는 product를 선택하고 Launch product를 클릭합니다.
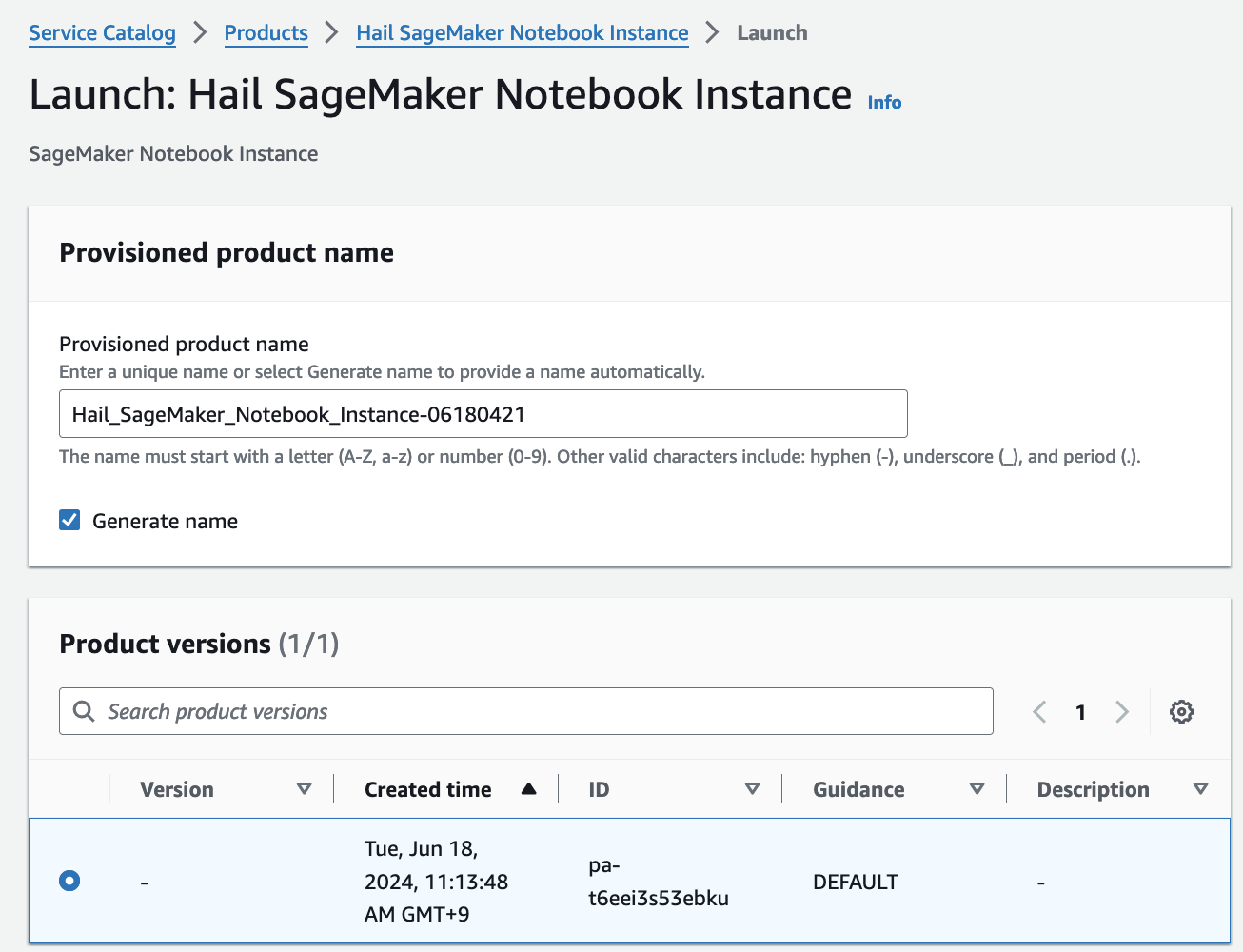
- Launch에 필요한 정보들을 기입합니다.
이름을 직접 입력하거나 Generate name을 클릭합니다.
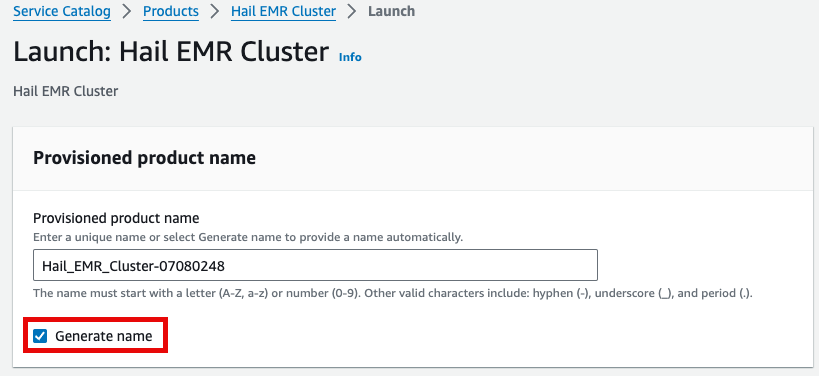
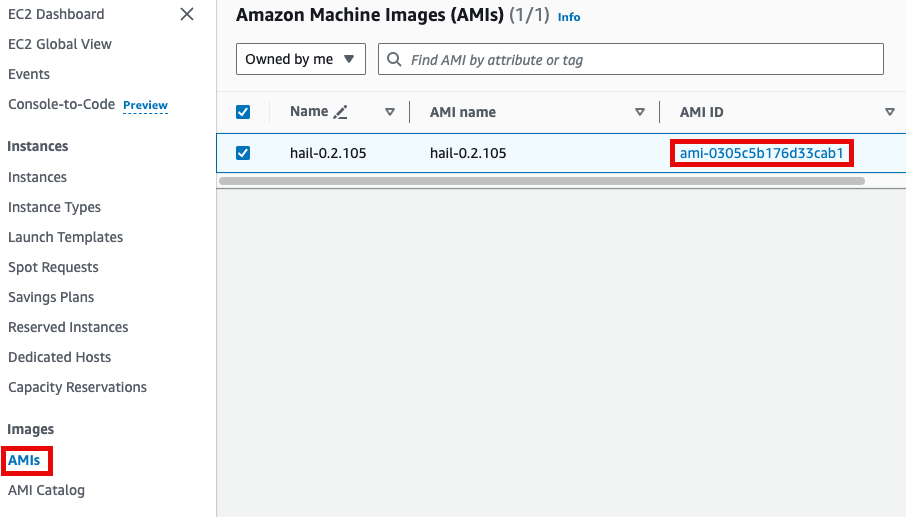
앞에서 만든 Hail AMI를 입력합니다. 이때 AMI ID는 AMI 메뉴에서 EC2 서비스 하위의 AMIs 항목에서 찾을 수 있습니다. (앞에서도 설명했던)
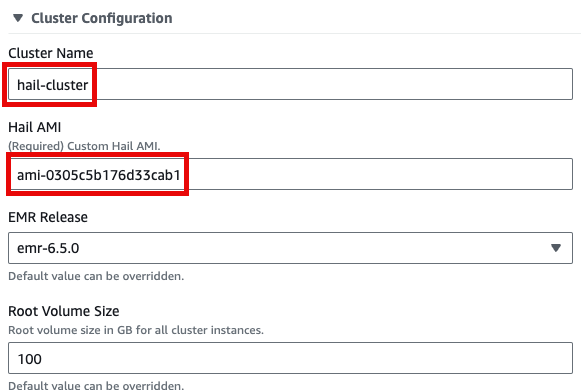
Cluster name을 입력하고 Hail AMI에 AMI ID를 입력한 뒤 다른 것은 모두 기본값을 사용할 수 있습니다.
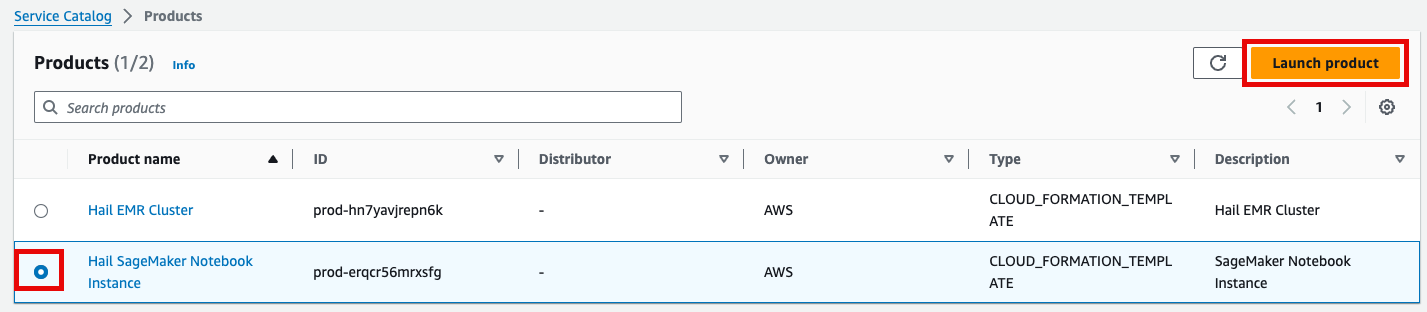
- 맨 아래의 Launch product를 클릭합니다.

SageMaker Notebook 실행
- Product 메뉴에서 마찬가지로 Launch product를 클릭합니다.
- Hail notebook을 위한 인스턴스의 이름을 입력하고 나머지는 모두 기본값입니다.
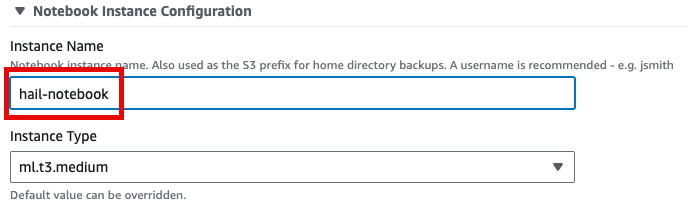
- 맨 아래의 Launch product를 클릭합니다.

참고로 제품(Product)의 실행 과정은 CloudFormation을 통해서도 확인할 수 있습니다.
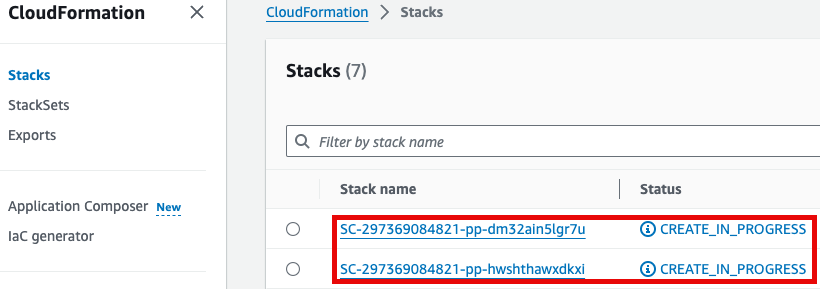
GWAS 실습 (Hail)
- 노트북을 실행합니다. 이 때 CloudFormation의 Outputs탭에서 url을 확인할 수 있습니다. 클릭하면 Amazon SageMaker의 해당 노트북 인스턴스로 자동 연결됩니다.
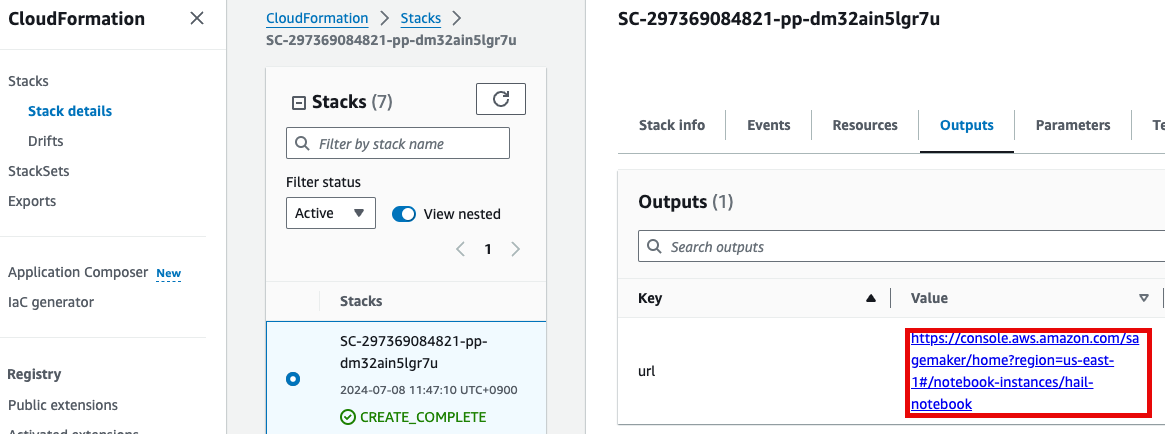
- Open JupyterLab을 클릭하여 노트북을 실행합니다.
- 이제 노트북에서 각각 2개의 노트북을 가지고 실습해봅니다.
- common-notebooks/plotting-tutorail.ipynb
- common-notebooks/GWAS-tutorial.ipynb
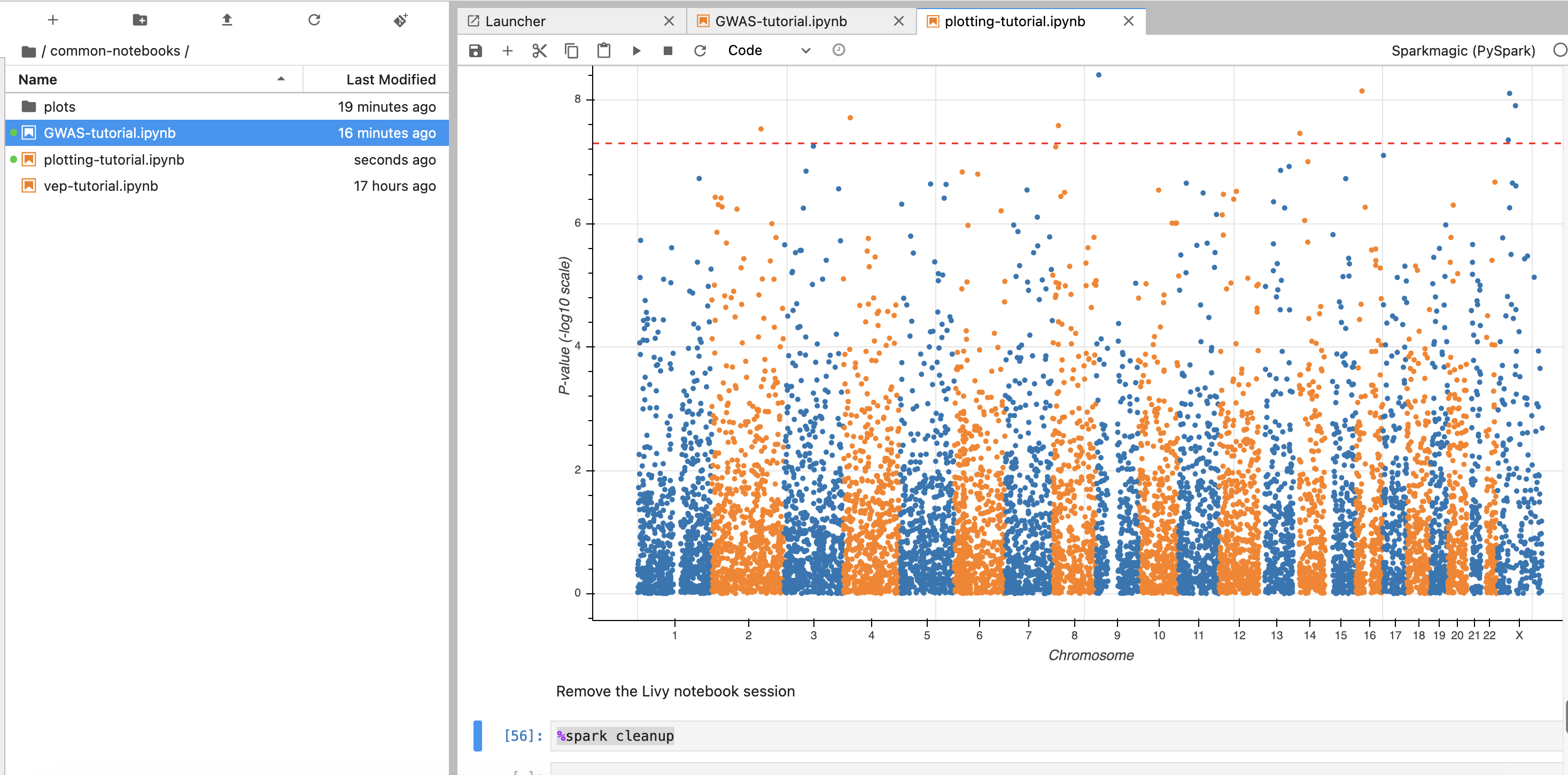
앞에서 만든 EMR 클러스터를 조회한 뒤 Cluster Name을 2번째 셀에서 수정해줍니다.
노트북 셀을 한번에 실행하기 위해서 시작하고자 하는 셀에 커서를 놓은 뒤 일괄 실행할 수 있습니다.
최종적으로 튜토리얼로 주어진 코드가 정상적으로 실행할 수 있었다면 아래와 같은 결과들을 확인할 수 있습니다.
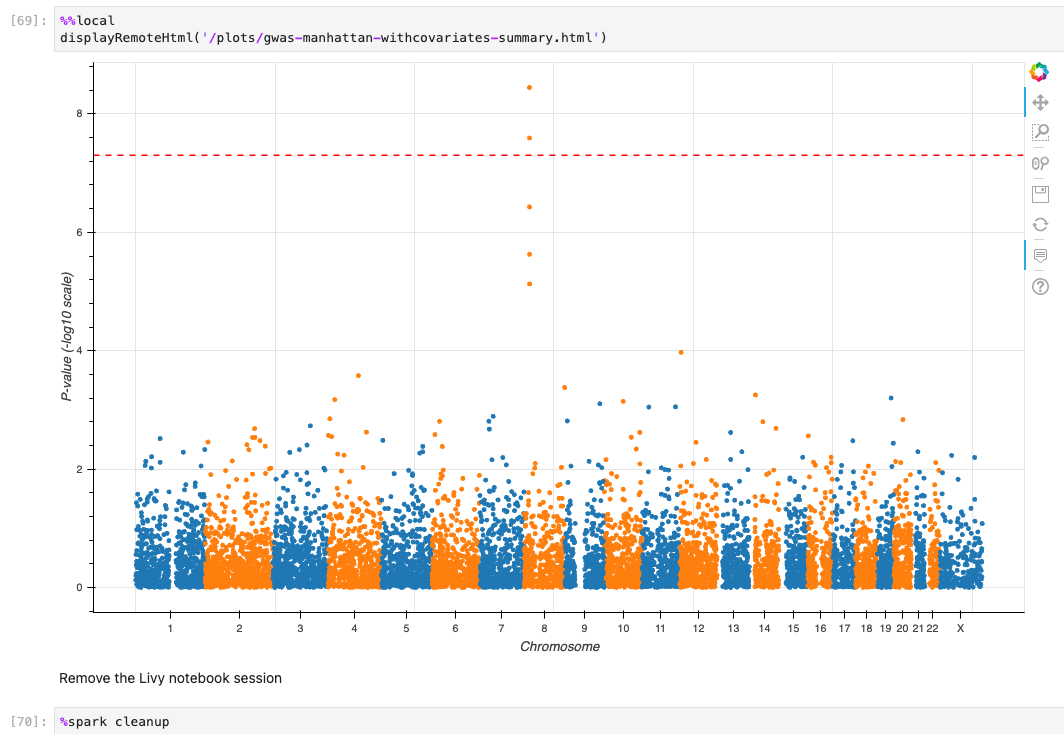
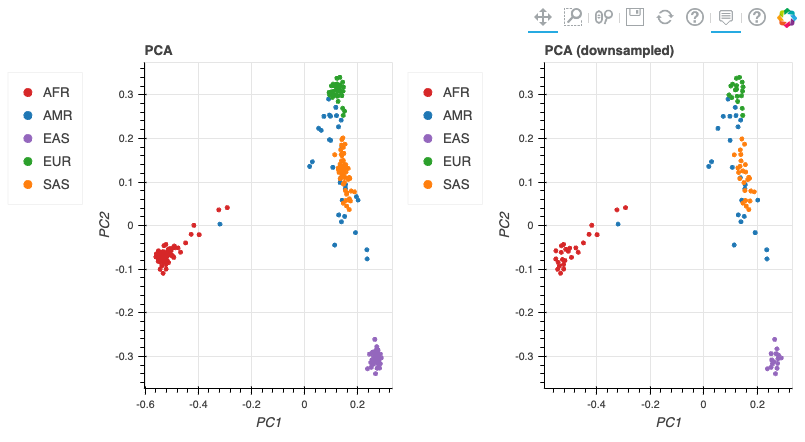
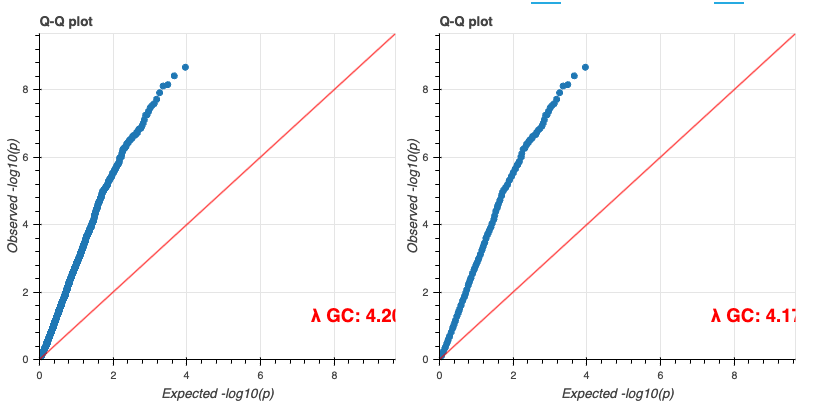
기타
VEP configuration
S3 버킷에서 해당 json파일 객체를 선택하고 Copy S3 URI를 클릭합니다.
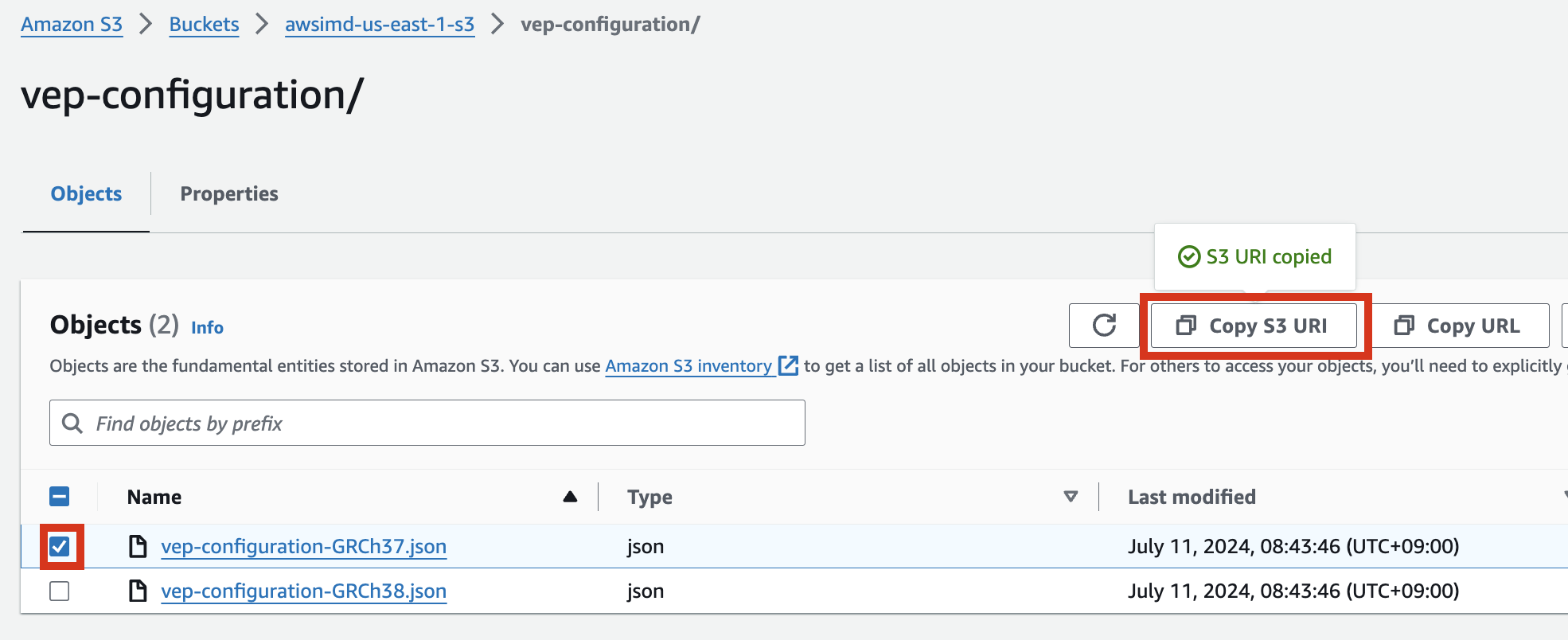
예시) vep-configuration-GRCh37.json
{
"command": [
"/opt/ensembl-vep/vep",
"--format", "vcf",
"--dir_plugins", "/opt/vep/plugins",
"--dir_cache", "/opt/vep/cache",
"--json",
"--everything",
"--allele_number",
"--no_stats",
"--cache", "--offline",
"--minimal",
"--assembly", "GRCh37",
"--plugin", "LoF,human_ancestor_fa:/opt/vep/loftee_data/human_ancestor.fa.gz,filter_position:0.05,min_intron_size:15,conservation_file:/opt/vep/loftee_data/phylocsf_gerp.sql,gerp_file:/opt/vep/loftee_data/GERP_scores.final.sorted.txt.gz",
"-o", "STDOUT"
],
"env": {
"PERL5LIB": "/opt/vep"
},
"vep_json_schema": "Struct{assembly_name:String,allele_string:String,ancestral:String,colocated_variants:Array[Struct{aa_allele:String,aa_maf:Float64,afr_allele:String,afr_maf:Float64,allele_string:String,amr_allele:String,amr_maf:Float64,clin_sig:Array[String],end:Int32,eas_allele:String,eas_maf:Float64,ea_allele:String,ea_maf:Float64,eur_allele:String,eur_maf:Float64,exac_adj_allele:String,exac_adj_maf:Float64,exac_allele:String,exac_afr_allele:String,exac_afr_maf:Float64,exac_amr_allele:String,exac_amr_maf:Float64,exac_eas_allele:String,exac_eas_maf:Float64,exac_fin_allele:String,exac_fin_maf:Float64,exac_maf:Float64,exac_nfe_allele:String,exac_nfe_maf:Float64,exac_oth_allele:String,exac_oth_maf:Float64,exac_sas_allele:String,exac_sas_maf:Float64,id:String,minor_allele:String,minor_allele_freq:Float64,phenotype_or_disease:Int32,pubmed:Array[Int32],sas_allele:String,sas_maf:Float64,somatic:Int32,start:Int32,strand:Int32}],context:String,end:Int32,id:String,input:String,intergenic_consequences:Array[Struct{allele_num:Int32,consequence_terms:Array[String],impact:String,minimised:Int32,variant_allele:String}],most_severe_consequence:String,motif_feature_consequences:Array[Struct{allele_num:Int32,consequence_terms:Array[String],high_inf_pos:String,impact:String,minimised:Int32,motif_feature_id:String,motif_name:String,motif_pos:Int32,motif_score_change:Float64,strand:Int32,variant_allele:String}],regulatory_feature_consequences:Array[Struct{allele_num:Int32,biotype:String,consequence_terms:Array[String],impact:String,minimised:Int32,regulatory_feature_id:String,variant_allele:String}],seq_region_name:String,start:Int32,strand:Int32,transcript_consequences:Array[Struct{allele_num:Int32,amino_acids:String,appris:String,biotype:String,canonical:Int32,ccds:String,cdna_start:Int32,cdna_end:Int32,cds_end:Int32,cds_start:Int32,codons:String,consequence_terms:Array[String],distance:Int32,domains:Array[Struct{db:String,name:String}],exon:String,gene_id:String,gene_pheno:Int32,gene_symbol:String,gene_symbol_source:String,hgnc_id:String,hgvsc:String,hgvsp:String,hgvs_offset:Int32,impact:String,intron:String,lof:String,lof_flags:String,lof_filter:String,lof_info:String,minimised:Int32,polyphen_prediction:String,polyphen_score:Float64,protein_end:Int32,protein_start:Int32,protein_id:String,sift_prediction:String,sift_score:Float64,strand:Int32,swissprot:String,transcript_id:String,trembl:String,tsl:Int32,uniparc:String,variant_allele:String}],variant_class:String}"
}
vep-tutorial 코드에서 아래 내용에서 위에서 복사한 S3 객체 URI로 수정하여 사용할 수 있습니다.
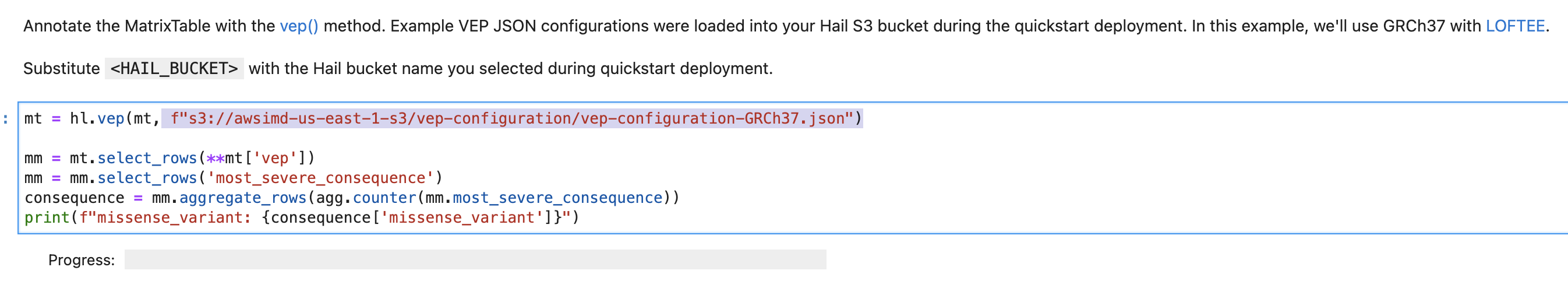
VEP 플러그인 설치
만일 VEP의 플러그인 설치에 변경사항 (추가 등)이 있다면 AMI를 다시 만들어야 합니다. 이 때 AMI를 만들 데 사용되는 VEP 설치에 관한 코드는 vep_install.sh 입니다. 해당 코드를 수정 후 다시 AMI를 빌드합니다.
다음을 참고하여 커스텀하게 Hail, VEP 툴을 설치 및 AMI를 빌드할 수 있습니다.
EMR 클러스터 EBS (HDFS) 동적 볼륨 늘리기
- 데이터가 클 경우 사전에 클러스터상에 구성된 볼륨의 용량이 부족할 수 있습니다. 아래 블로그 내용을 참고하여, EBS 볼륨의 부족분을 동적으로 늘릴 수 있습니다.
https://aws.amazon.com/ko/blogs/big-data/dynamically-scale-up-storage-on-amazon-emr-clusters/

No Comments